Determinantes da resposta aos inibidores de CDK4/6 no mundo real
Abstrato
Apesar do uso generalizado e de um mecanismo de ação conhecido para os inibidores de CDK4/6 em combinação com a terapia endócrina, as características da evolução da doença e os determinantes da resposta terapêutica no cenário do mundo real permanecem obscuros. Aqui, uma coorte de pacientes tratados com regimes combinados de tratamento padrão foi utilizada para explorar características da doença e determinantes da sobrevida livre de progressão (PFS) e da sobrevida global (SG). Nesta coorte de 280 pacientes, >90% dos pacientes foram tratados com palbociclib em combinação com um inibidor da aromatase (IA) ou fulvestrant (FUL). A maioria desses pacientes apresentava escores modificados de Scarff-Bloom-Richardson (SBR) e imuno-histoquímica de ER, HER2 e PR. Tanto o escore SBR quanto a falta de expressão de PR foram associados a PFS mais curta em pacientes tratados com combinações de IA e permaneceram significativos em análises multivariadas (HR = 3,86, p = 0,008). As análises de expressão gênica indicaram mudanças substanciais no ciclo celular e na sinalização do receptor de estrogênio durante o tratamento. Além disso, a subtipagem baseada na expressão gênica indicou que os subtipos predominantes mudaram com o tratamento e a progressão. Os subtipos luminal B, HER2 e basal exibiram PFS mais curto em combinações de inibidores de CDK4/6 quando avaliados nas biópsias pré-tratamento; no entanto, eles não estavam associados ao sistema operacional. Utilizando abordagens imparciais, os conjuntos de genes associados ao ciclo celular foram fortemente associados a PFS mais curtos em biópsias pré-tratamento, independentemente da terapia endócrina. Os conjuntos de genes de sinalização do receptor de estrogênio foram associados a uma PFS mais longa, particularmente na coorte tratada com IA. Juntos, estes dados sugerem que existem características patológicas e biológicas distintas do câncer de mama HR +/HER2− associadas à resposta aos inibidores de CDK4/6. Número de registro de ensaio clínico: NCT04526587.
Conteúdo semelhante sendo visualizado por outras pessoas
Introdução
O câncer de mama com receptor hormonal positivo e HER2 negativo (HR + / HER2−) representa uma das doenças malignas mais prevalentes no mundo ocidental. O tratamento da doença localizada envolve cirurgia, quimioterapia (se indicada) e radiação, seguida de terapia endócrina no cenário adjuvante 1 , 2 . Assinaturas de expressão gênica foram desenvolvidas para determinar o risco de recorrência da doença em estágio inicial e para elucidar o benefício da quimioterapia ou do tratamento prolongado com terapia endócrina 3 , 4 , 5 . Estas abordagens de precisão são eficazes; entretanto, um número significativo de pacientes desenvolve doença metastática recorrente ou apresenta doença metastática de novo. O câncer de mama metastático recorrente HR+/HER2− pode se desenvolver ao longo de muitos anos e representa um risco contínuo 6 , 7 .
O tratamento do câncer de mama metastático HR+/HER2− evoluiu 1 . Historicamente, em mulheres pós-menopáusicas, a terapia endócrina, tanto inibidores da aromatase (IA) quanto degradadores seletivos de estrogênio (SERDS), eram prescritos para o tratamento da doença metastática. Estas terapias podem prolongar a sobrevivência livre de progressão (PFS), mas as respostas são de duração limitada e os tratamentos não são curativos. O PFS para o letrozol IA padrão de tratamento é de aproximadamente 14–16 meses, enquanto o PFS do SERD fulvestrant de tratamento padrão é de aproximadamente 6–8 meses 8 . Vários ensaios clínicos questionaram a utilização de agentes direcionados em conjunto com a terapia endócrina para aumentar a durabilidade da resposta 9 . Estes ensaios aleatorizados apoiaram a utilização de inibidores de CDK4/6 para limitar a progressão da doença e, em certos contextos, produzir um aumento na sobrevivência global (SG) 8 , 10 , 11 , 12 , 13 , 14 , 15 , 16 . Atualmente, três inibidores de CDK4/6 são aprovados pela FDA para o tratamento de câncer de mama metastático HR+/HER2−. Estas terapias geralmente duplicam a PFS da terapia endócrina sozinha; no entanto, há um pequeno grupo de pacientes (15–25%) que progride rapidamente independentemente do inibidor de CDK4/6, sugerindo a existência de tumores que são intrinsecamente resistentes ao inibidor de CDK4/6 e ao tratamento combinado de terapia endócrina.
Estudos pré-clínicos forneceram informações sobre os determinantes da resposta à inibição de CDK4/6. Foi demonstrado que modelos de câncer de mama deficientes em RB são resistentes aos inibidores de CDK4/6 17 , 18 . Estes modelos geralmente exibem expressão elevada de p16 Ink4a concomitante com a interrupção da função RB 19 . Embora este estado pareça ser relativamente comum no cancro da mama triplo negativo, a perda de RB é pouco frequente no cancro da mama HR+/HER2− 20 , 21 , 22 . No entanto, análises diretas do status de RB indicaram PFS mais curta em pacientes com perda de RB em comparação com homólogos do tipo selvagem 20 , 22 . Vários mecanismos diferentes surgiram relacionados à resistência, incluindo a desregulação da Ciclina E, CDK6, via RAS, expressão AMBRA1, MYC e via HIPPO 21 , 23 , 24 , 25 , 26 , 27 . Notavelmente, cada um destes eventos genéticos compromete, em última análise, a actividade do inibidor de CDK4/6 na indução de uma potente paragem do ciclo celular.
Os marcadores utilizados para prever a resposta aos inibidores de CDK4/6 ainda não foram desenvolvidos para aplicação clínica 22 , 28 . No contexto da terapia endócrina adjuvante, vários painéis de expressão gênica são comumente utilizados. 3 , 29 , 30 . Estas assinaturas geralmente definem o risco de recorrência, o benefício correspondente da quimioterapia e/ou a necessidade de tratamento mais prolongado com terapia endócrina. Curiosamente, a maioria destas assinaturas abriga genes associados à proliferação que são regulados pela via RB 31 , 32 . Por exemplo, o Oncotype Dx é composto por módulos que interrogam amplamente a sinalização do receptor de estrogênio e o status de proliferação; da mesma forma, a subtipagem intrínseca do PAM50 utiliza genes associados à proliferação para diferenciar A e B luminal 5 , 32 . Portanto, várias assinaturas diferentes que mostram as aberrações da via RB são prognósticas em coortes de câncer de mama HR+/HER2− 33 , 34 , 35 , 36 , 37 . É importante ressaltar que, devido à amostragem rotineira de câncer de mama para avaliar marcadores (isto é, receptor de estrogênio e HER2), a maioria das estratégias de biomarcadores permanece baseada em amostras de tecido.
Devido ao mecanismo de ação do inibidor de CDK4/6, existem vários determinantes suspeitos de resposta, que incluiriam perda de RB e superexpressão de p16 Ink4a . Embora a perda de RB pareça estar associada à PFS curta com inibidores de CDK4/6 22 , é um evento raro no câncer de mama metastático sem inibidor de CDK4/6. Da mesma forma, a expressão do gene da Ciclina E tem sido associada a uma PFS mais curta 23 . Uma variedade de análises de biomarcadores de ensaios clínicos randomizados identificaram supostos determinantes para a duração da PFS 26 , 38 , 39 , 40 . No entanto, as análises das características da resposta no ambiente de tratamento padrão do mundo real, incorporando marcadores patológicos e histológicos padrão, ainda não foram avaliadas. Aqui, examinamos uma coorte de pacientes tratados com inibidores de CDK4/6 para delinear características de evolução durante o tratamento como potenciais determinantes da PFS que poderiam ser aplicáveis à prática padrão.
Resultados
Coorte de pacientes e análise de marcadores histológicos e patológicos padrão
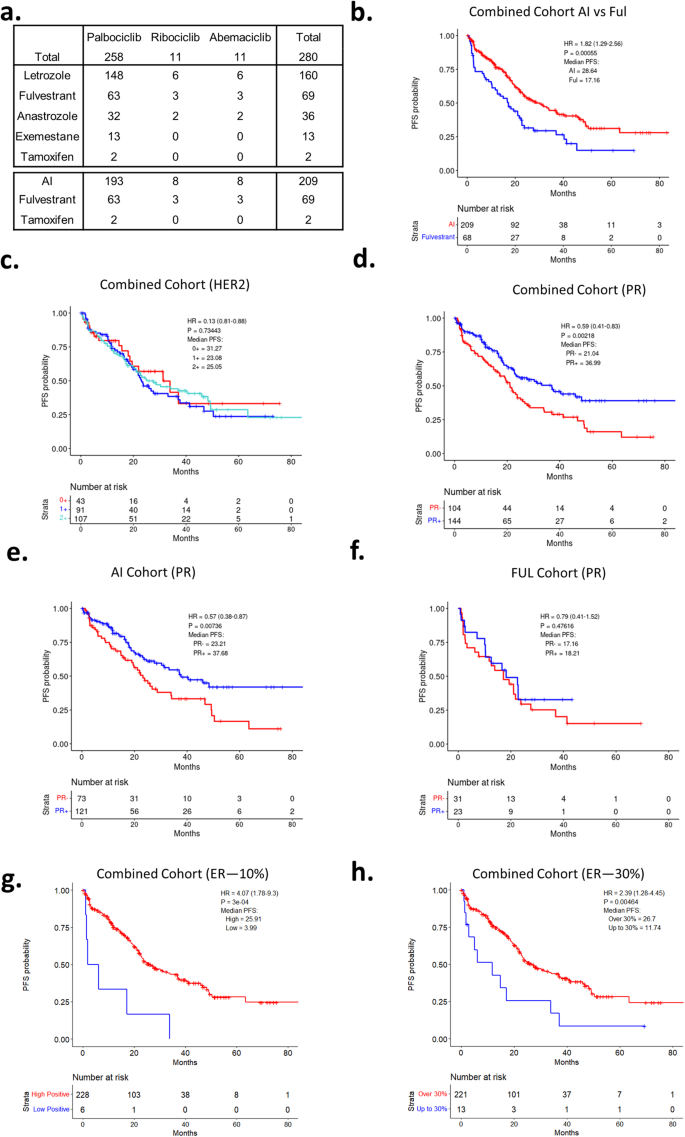
A fim de definir os determinantes da resposta aos inibidores de CDK4/6 utilizados no ambiente de tratamento padrão, foi desenvolvido um estudo aprovado pelo IRB (NCT04526587) em paralelo com a revisão retrospectiva de prontuários. Mais de 3.500 pacientes foram selecionados conforme resumido no diagrama CONSORT (Figura 1 Complementar ). Um total de 280 pacientes foram avaliados em dezembro de 2022 (Tabela 1 ). Informações clínicas e patológicas detalhadas foram obtidas por revisão de prontuários e resumidas em um banco de dados REDCap. Nesta população de pacientes, a maioria (92%) foi tratada com palbociclib juntamente com um inibidor da aromatase (AI) ou fulvestrant (FUL), conforme resumido (Fig. 1a ). Nesta coorte, a sobrevida livre de progressão (PFS) para pacientes tratados com as combinações AI ou FUL foi de 28,6 e 17,2 meses, respectivamente (Fig. 1b ), o que é comparável ao observado em ensaios clínicos randomizados 8 , 41 , 42 . Entre as variáveis clínicas, o envolvimento visceral, a terapia endócrina prévia e a doença recorrente foram associados a uma PFS mais curta (Figura 1 Complementar ), consistente com outros estudos. Nesta coorte, a OS foi determinada a partir do início do tratamento ou do ponto de progressão. Desde o início do tratamento, o grupo tratado com IA apresentou uma OS mais longa (Figura 1 Complementar ); no entanto, ao utilizar o ponto de progressão na terapia baseada no inibidor de CDK4/6 como ponto de partida, a OS para cada grupo de tratamento foi verdadeiramente idêntica (OS mediana ~19 meses) (Figura 1 Complementar ) . Como seria de esperar, nesta coorte a terapia pós-progressão foi altamente variada (Figura 1 Complementar ).
a Tabela que resume a combinação de terapia CDK e terapia hormonal por paciente. b Análise de Kaplan-Meier da sobrevida livre de progressão comparando combinações de CDK4/6i com fulvestranto versus IA. p = 0,00055 por log rank. c Análise de Kaplan-Meier da sobrevida livre de progressão comparando o subtipo HER2 resultante por IHQ em todos os pacientes. p = 0,73 por log rank. d Análise de Kaplan-Meier da sobrevida livre de progressão comparando o status de RP em todos os pacientes. p = 0,0022 por log rank. e Análise de Kaplan-Meier da sobrevida livre de progressão comparando o status de RP para pacientes em uso de IA. p = 0,0074 por log rank. f Análise de Kaplan-Meier da sobrevida livre de progressão comparando o status de RP para pacientes em uso de fulvestrant. p = 0,48 por log rank. g Análise de Kaplan-Meier da sobrevida livre de progressão comparando o status do RE dividido com intensidade de coloração de 10% em todos os pacientes. p = 3e −4 por log rank. h Análise de Kaplan-Meier da sobrevida livre de progressão comparando o status do RE dividido com intensidade de coloração de 30% em todos os pacientes. p = 0,005 por log rank.
Para determinar se algum marcador patológico padrão estava associado à PFS, avaliamos as expressões do receptor de progesterona (PR) e HER2. Descobrimos que o nível de HER2 determinado pela pontuação IHC (0, 1+ ou 2+/FISH não amplificado) não estava associado à PFS na coorte combinada (Fig. 1c ) ou na análise de subgrupo de IA ou Pacientes tratados com FUL (Figura 2 suplementar ). Em contraste, o status de RP foi associado à PFS, com status negativo/baixo de RP denotando uma PFS significativamente mais curta na coorte combinada (Fig. 1d ). Nas análises de subgrupos, a ausência de RP foi especificamente associada à PFS nos pacientes tratados com IA, mas não nos pacientes tratados com FUL (Fig. 1e, f ). Como a expressão do PR é um substituto da atividade do ER, também avaliamos a influência do status do ER na PFS. Embora todos os casos sejam positivos para RE, um pequeno subconjunto apresenta baixa positividade (<10%). Este estado foi associado a PFS mais curto (Fig. 1g ), da mesma forma, níveis baixos/médios de coloração de ER (<30%) foram associados a PFS encurtado (Fig. 1h ).
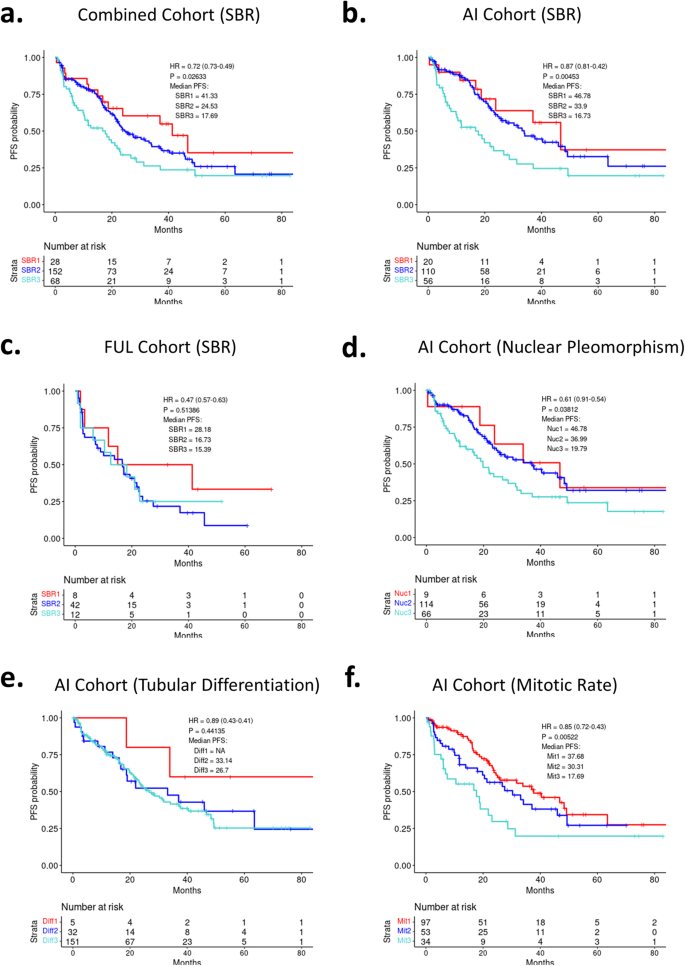
A maioria dos pacientes ( n = 248) teve o grau histológico do tumor atribuído usando a modificação de Nottingham da pontuação de Scarff-Bloom-Richardson (SBR). Pontuações SBR mais altas foram associadas a PFS mais curta no contexto da coorte combinada (Fig. 2a ), bem como no subgrupo tratado com IA (Fig. 2b ), mas não no grupo tratado com FUL (Fig. 2c ). Como diferentes amostras de tecido (ressecção e biópsia) foram utilizadas para a determinação da SBR, os procedimentos de obtenção de tecido também foram avaliados e não alteraram significativamente a associação com a PFS (não mostrado). SBR é composto por graus de diferenciação tubular, pleomorfismo nuclear e taxa mitótica; cada componente individual foi analisado na coorte combinada (Fig. Complementar 3 ), bem como no grupo tratado com IA (Fig. 2d-f ). Nesse contexto, a taxa mitótica e os graus de pleomorfismo nuclear foram associados ao desfecho, mas não à diferenciação tubular. As variáveis do escore SBR não foram associadas à PFS no subgrupo FUL (Figura 3 Complementar ). As análises univariadas dos marcadores patológicos e histológicos estão resumidas na Tabela 2 . Empregando um modelo multivariado que incorporou os marcadores significativos, em conjunto com a doença metastática visceral e a terapia endócrina prévia, a PR e a SBR mantiveram a significância (Tabela 2 ).
uma análise de Kaplan-Meier da sobrevida livre de progressão comparando o escore SBR geral em todos os pacientes. p = 0,026 por log rank. b Análise de Kaplan-Meier da sobrevida livre de progressão comparando a pontuação geral do SBR entre pacientes em uso de IA. p = 0,0045 por log rank. c Análise de Kaplan-Meier da sobrevida livre de progressão comparando o escore SBR geral entre pacientes que tomam fulvestrant. p = 0,51 por log rank. d Análise de Kaplan-Meier da sobrevida livre de progressão comparando o Pleomorfismo Nuclear entre pacientes em uso de IA. p = 0,038 por log rank. e Análise de Kaplan-Meier da sobrevida livre de progressão comparando a Diferenciação Tubular entre pacientes em uso de IA. p = 0,44 por log rank. f Análise de Kaplan-Meier da sobrevida livre de progressão comparando a taxa mitótica entre pacientes em uso de IA. p = 0,0052 por log rank.
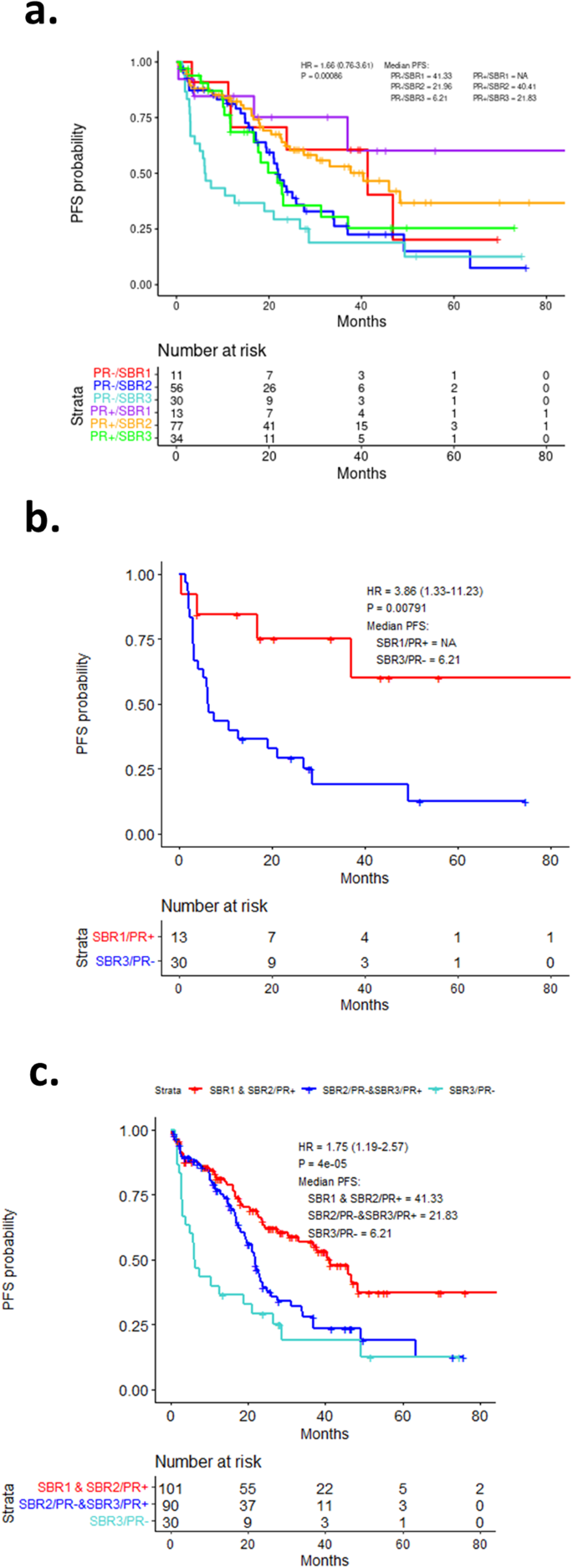
Esses achados sugerem que o status de PR e SBR poderia ser utilizado em combinação para definir tumores com PFS longo versus curto previsto. Na coorte combinada, a combinação do status PR e SBR foi significativamente associada à PFS (Fig. 3a ). Em particular, a combinação de status SBR3 e PR negativo foi associada a uma PFS excessivamente curta, enquanto ser SBR1 e PR positivo foi associado a uma maior duração de PFS (Fig. 3b ). Outras combinações de SBR e PR foram associadas à PFS intermediária (Fig. 3c ). Como esperado, as pontuações combinadas de SBR e PR foram particularmente relevantes na coorte tratada com inibidor de AI + CDK4/6 (Figura 4 suplementar ).
uma análise de Kaplan-Meier da sobrevida livre de progressão comparando o status de PR e a pontuação geral do SBR em todos os pacientes. p = 0,00086 por log rank. b Análise de Kaplan-Meier da sobrevida livre de progressão comparando os extremos PR/SBR em todos os pacientes. p = 0,0079 por log rank. c Análise de Kaplan-Meier da sobrevida livre de progressão comparando vários subgrupos PR/SBR em todos os pacientes. p < 0,0001 por log rank.
Evolução tumoral
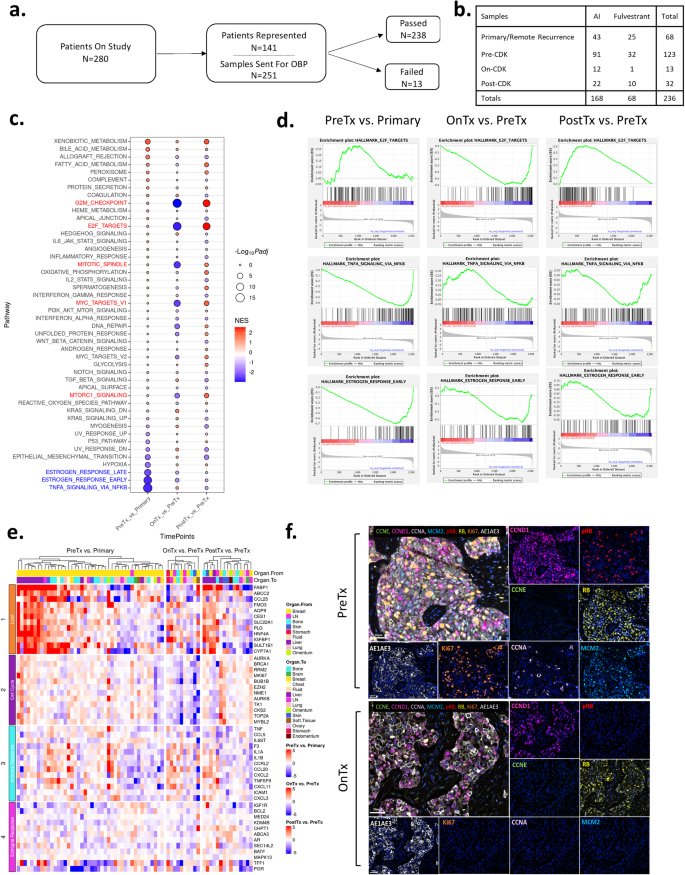
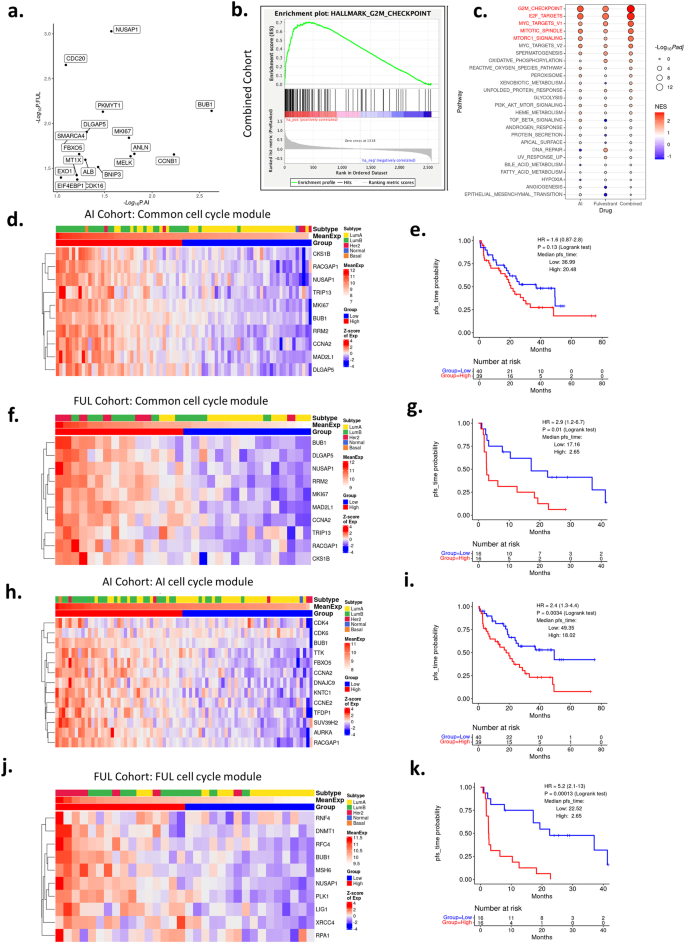
Tecido tumoral foi obtido no tratamento clínico padrão de 141 pacientes (por exemplo, de biópsias clinicamente obrigatórias). Uma vez que múltiplos tecidos podem estar disponíveis para um determinado caso clínico, um total de 251 amostras foram empregadas para análises de expressão gênica direcionadas usando o HTG Oncology Biomarker Panel de 2.549 genes, dos quais 238 amostras passaram no controle de qualidade (Fig. 4a e Fig. Complementar 1) . ). Esses tecidos foram subdivididos com base no tratamento (foram excluídos 2 tecidos de tumores tratados com tamoxifeno), bem como no estágio clínico/ponto no tempo em que o tecido foi obtido (Fig. 4b ). As amostras de recorrência primária e remota representam tecidos que foram obtidos significativamente antes de receber uma terapia baseada em inibidor de CDK4/6 (média = 78,2 meses), os tecidos pré-tratamento foram obtidos antes do início do tratamento (média = 3,6 meses), nos casos de tratamento foram enquanto o paciente estava recebendo terapia, enquanto a pós-progressão ocorreu após progressão clínica na terapia baseada em inibidor de CDK4/6 (Fig. 4b ). Para determinar características associadas à doença através do tratamento, amostras pareadas do mesmo paciente foram utilizadas para definir genes diferencialmente expressos entre recorrência primária/remota e biópsias pré-tratamento, bem como durante e pós-tratamento. A análise de enriquecimento de conjunto de genes classificados (GSEA) foi implantada para investigar características biológicas associadas a cada etapa do cenário da doença (Fig. 4c ). Notavelmente, com a progressão para doença metastática, há diminuições na sinalização da resposta ao estrogênio e na sinalização do TNF via NF Kappa B (NFKB) (denotado em azul). Há um aumento em termos relacionados ao metabolismo de xenobióticos e ácidos biliares e fraca indução de assinaturas relacionadas ao ciclo celular (indicadas em vermelho) (Fig. 4c, d ). No tratamento, há uma inibição profunda das assinaturas do ciclo celular e uma indução modesta de genes de sinalização do TNF (Fig. 4c, d ). Por último, com a progressão, há maior desregulação do ciclo celular e diminuição da sinalização do receptor de estrogênio e dos conjuntos de genes de sinalização do TNF (Fig. 4c, d ). Para explorar a diferença de nível de gene/assinatura, mapas de calor foram usados para avaliar mudanças na expressão gênica em relação ao local metastático e outras características da doença (Fig. 4e). Estas análises indicaram que o metabolismo xenobiótico e conjuntos de genes semelhantes estão associados à metástase hepática, indicando a importância de ter informações clínicas detalhadas para definir a base de assinaturas seletivas nos dados. As alterações no ciclo celular, na sinalização do TNF e na sinalização do estrogênio foram características relativamente gerais e não restritas a um local metastático específico. Para avaliar as alterações durante o tratamento e pós-progressão, elas foram comparadas com dados do NeoPalAna, onde os pacientes foram tratados com palbociclib neoadjuvante em combinação com anastrozol 43 . Estes dados ilustraram fluxos semelhantes na expressão genética no tratamento que são revertidos após a progressão (Figura 5 suplementar ). Por último, para avaliar os efeitos do tratamento relacionados às proteínas do ciclo celular, utilizamos coloração de imunofluorescência multiespectral em um subconjunto de amostras pareadas (Fig. 4f e Fig. Complementar 5 ). Esses dados mostraram a perda seletiva de RB fosforilado com o tratamento, enquanto a proteína total foi retida consistente com a ação dos inibidores de CDK4/6 44 . Os alvos a jusante MCM2, Cyclin A e Ki67 foram suprimidos, de acordo com o efeito na expressão destes genes no tratamento. A ciclina D1, que se sabe ser retida no contexto do tratamento com inibidor de CDK4/6 44 , estava presente tanto nas amostras pré como durante o tratamento.
Fluxograma representativo das amostras do estudo enviadas para OBP. b Tabela que resume o ponto temporal do tecido para todas as amostras de IA e fulvestrant que passaram no OBP. c Conjuntos de genes enriquecidos para genes expressos diferencialmente entre diferentes pontos no tempo usando amostras pareadas dos mesmos pacientes. As alterações dobradas de Log2 são calculadas usando DESeq2 na configuração do modo emparelhado e usadas como lista de genes classificados para entrada no GSEA. A pontuação de enriquecimento normalizada (NES) juntamente com os valores de p associados são usados para gerar este gráfico de bolhas. d Gráficos de enriquecimento GSEA para conjuntos de genes selecionados em cada uma das três comparações de pontos no tempo. e Mapa de calor representando flutuações de expressão gênica para genes selecionados em diferentes grupos funcionais durante o curso da terapia. f Imagem de imunofluorescência multiespectral de uma amostra pareada de pré-tratamento e durante o tratamento.
Subtipos intrínsecos e associação com PFS e OS
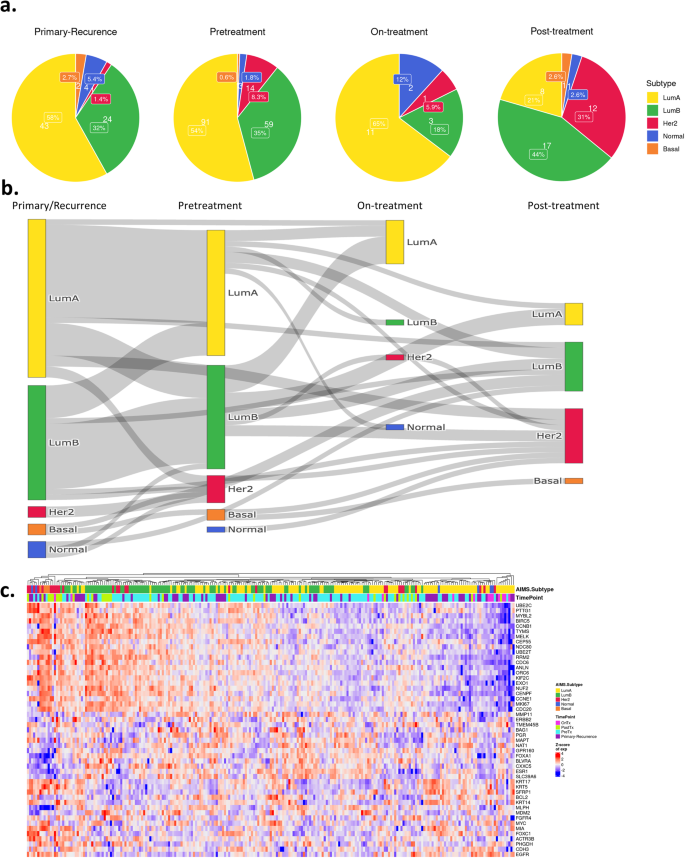
O pacote Atribuição Absoluta de Subtipos Intrínsecos de Câncer de Mama (AIMS) 45 foi utilizado para atribuir o subtipo de cada amostra. Como esperado, a maioria dos tumores foi classificada como luminal A ou B. O tumor primário/recorrências remotas antes do início da terapia com CDK4/6 exibiram uma frequência semelhante de subtipos luminais A e B, conforme observado no cenário metastático pré-tratamento (Fig. 5a ). Embora o número de biópsias em tratamento tenha sido limitado, elas foram dominadas pelo subtipo luminal A/normal (Fig. 5a ). Por último, na doença pós-progressão, o luminal A foi observado na minoria dos tumores (Fig. 5a ). Estes dados sugerem que durante o tratamento existem flutuações dos subtipos, o que é ilustrado na análise de Sankey mostrando as trajetórias entre amostras emparelhadas (Fig. 5b ). Para avaliar melhor as alterações ao nível do gene, a assinatura PAM50 foi avaliada em todas as amostras (Fig. 5c ). Estas análises mostram a associação geral de amostras pós-progressão com alta expressão de genes do ciclo celular (por exemplo, PTTG1, UBE2C e CCNB1) e menor expressão de genes associados à sinalização de estrogênio (por exemplo, PGR, ESR1 e FOXA1).
a Gráficos circulares que comparam os subtipos de cancro da mama ao longo dos pontos temporais dos tecidos, conforme previsto pela Atribuição Absoluta do Subtipo Molecular Intrínseco do Cancro da Mama (AIMS). b Gráfico de Sankey comparando os subtipos de câncer de mama entre os pontos temporais dos tecidos, conforme previsto pelo AIMS. c Mapa de calor mostrando ativação/supressão de genes no conjunto de genes PAM50. Os subtipos previstos pela AIMS mostraram forte associação com os programas de expressão gênica dos pacientes que utilizam esse biomarcador.
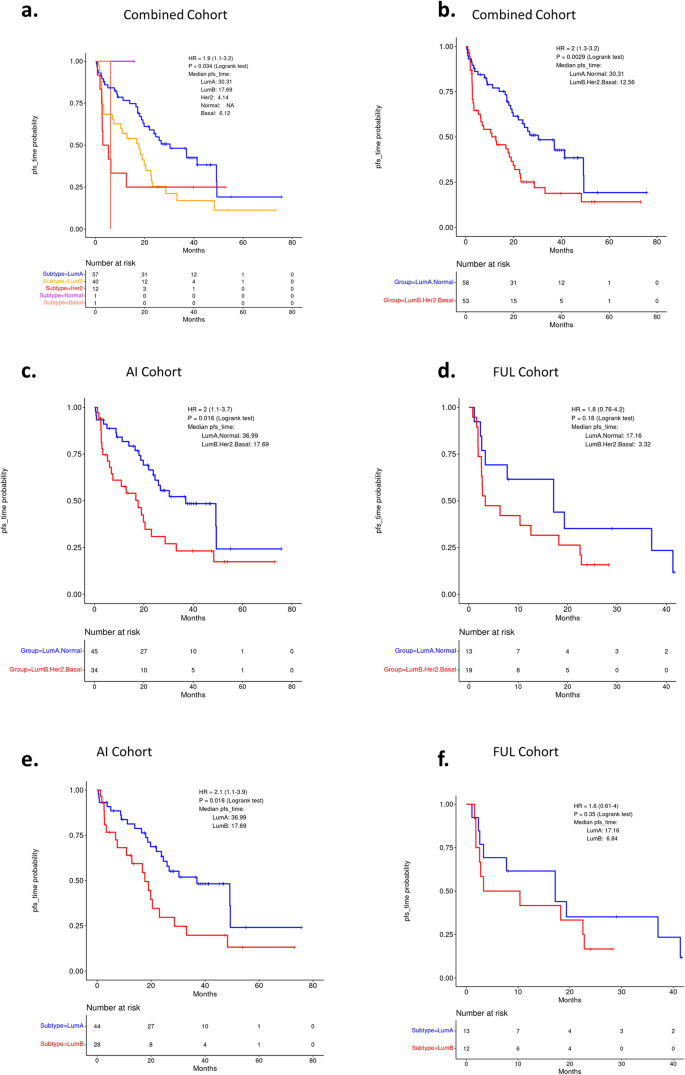
Na análise da relação dos subtipos com a PFS, inicialmente foram empregadas amostras de pré-tratamento. Neste cenário, os subtipos AIMS foram associados à PFS (Fig. 6a ). A separação de subtipos de prognóstico historicamente ruim (luminal B, basal e HER2) de subtipos de melhor prognóstico (luminal A e normal) ilustrou a associação com PFS na coorte completa (Fig. 6b ). Esta estratificação foi significativa em subgrupos de pacientes tratados com IA, mas não com FUL, embora isso possa ser devido ao número relativamente pequeno de casos no subgrupo FUL (Fig. 6c, d ). Da mesma forma, a estratificação do luminal A vs luminal B foi significativa apenas no grupo tratado com IA (Fig. 6e, f ). Quando aplicados às amostras de recorrência remota/tumor primário, os subtipos AIMS apenas tenderam à significância, apoiando a importância potencial da avaliação das amostras metastáticas pré-tratamento (Figura 6 suplementar ). Curiosamente, ao avaliar a duração da OS, os subtipos foram apenas modestamente associados à sobrevivência, embora os subtipos não luminais invariavelmente associados a resultados piores (Figura 6 suplementar ).
Testes para associação de subtipos intrínsecos previstos pela AIMS com PFS. a Coortes combinadas (tratadas com IA e fulvestrant) para subtipos individuais. p = 0,034 pelo teste log rank. b Coorte combinada nos grupos luminal A e normal, luminal B, Her2 e basal combinados. p = 0,0029 pelo teste de log rank. c , d coortes tratadas com AI ou fulvestrant separadamente em subtipos combinados. p = 0,016 e p = 0,18, respectivamente, pelo teste log rank. e , f coortes tratadas com AI ou fulvestrant separadamente entre os subtipos luminal A e luminal B. p = 0,018 e p = 0,35, respectivamente, pelo teste log rank.
Genes reguladores de CDK e duração da PFS
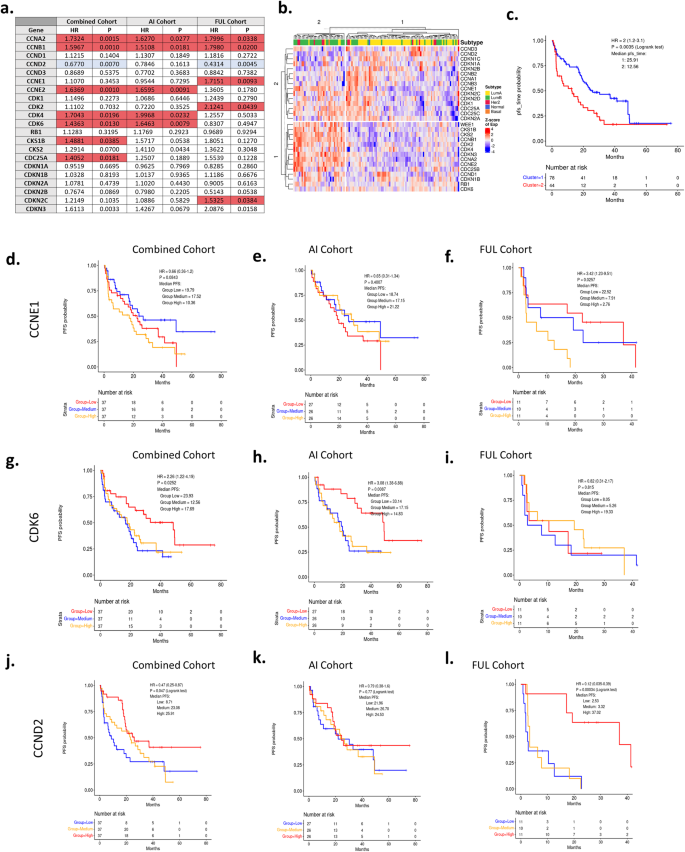
A partir de estudos pré-clínicos, foi sugerido que vários genes associados à regulação de CDK desempenham um papel na resposta aos inibidores de CDK4/6. Aqui avaliamos ciclinas e CDKs relacionadas ao ciclo celular e reguladores-chave (por exemplo, CDKN1B e CKS1B) de uma forma relativamente imparcial. Um subconjunto destes genes foi associado a uma PFS mais curta (vermelho), com apenas um gene associado a uma PFS mais longa (azul) (Fig. 7a ). Curiosamente, vários genes suspeitos de estarem associados à resposta (por exemplo, CCND1 e RB1) não foram associados à PFS (Figura 7 Complementar ). O agrupamento hierárquico não supervisionado ilustrou que existe um grau de co-regulação dos genes do ciclo celular, em que a alta expressão de muitos genes do ciclo celular está associada a PFS mais curto (Fig. 7b, c ). O comportamento de CCNE1 e CDK6 é mostrado pelos gráficos de Kaplan-Meier (KM) na coorte combinada e nas subcoortes seletivas de tratamento (Fig. 7d-i ). O único gene identificado que se associa a uma maior duração da PFS é o CCND2 (Fig. 7j-l ), que está inversamente correlacionado com muitos dos outros genes regulados pelo ciclo celular e é responsivo à sinalização interferon/NF Kappa B 46 , 47 .
a Tabela que resume a associação de PFS com os principais genes do ciclo celular em coortes de pacientes. b Mapa de calor mostrando a expressão desses genes em biópsias pré-tratamento. Este agrupamento não supervisionado é capaz de estratificar pacientes luminal A de outros subtipos (principalmente luminal B). c Gráfico de Kaplan-Meier mostrando diferença de sobrevida livre de progressão entre os dois grupos estratificados por agrupamento não supervisionado. p = 0,0035 pelo teste de log rank. d – l Teste do poder de genes únicos na estratificação de pacientes para sobrevida livre de progressão usando dados de expressão gênica de biópsias pré-tratamento. Para CCNE1 nas coortes combinadas, AI e FUL, p = 0,0843, p = 0,4007 e p = 0,0257 pelo teste log rank, respectivamente. Para CDK6 nas coortes combinadas, AI e FUL, p = 0,0252, p = 0,0087 e p = 0,815 pelo teste log rank, respectivamente. Para CCND2 nas coortes combinadas, AI e FUL, p = 0,047, p = 0,77 e p < 0,001 pelo teste log rank, respectivamente.
Características biológicas associadas ao PFS
Os dados com subtipagem PR, SBR e AIMS sugerem que existem características intrínsecas associadas à PFS em tumores tratados com inibidores de CDK4/6 e terapia endócrina. Para explorar características associadas à duração da SLP, a coorte foi dividida de acordo com a terapia endócrina (AI ou FUL). Para cada tipo de terapia, foi realizada uma análise de regressão de risco proporcional univariada de Cox para definir genes que foram independentemente associados (em termos de taxas de risco e valores de p ) com sensibilidade ou resistência às terapias (Conjunto de Dados Suplementares 1 ). Como mostrado na Fig. 8a , uma coleção de genes foi associada a PFS mais curta em ambos os grupos de tratamento AI e FUL. Para determinar as características biológicas associadas à sensibilidade ou resistência, empregamos a Análise de Enriquecimento de Conjunto de Genes (GSEA) classificada na coorte combinada 48 . Nesta análise, os genes são classificados em taxas de risco e testados quanto ao enriquecimento para os conjuntos de genes característicos do Banco de Dados de Assinaturas Moleculares (MSigDB) 48 , 49 na extremidade superior e inferior do espectro de HR. Estas análises indicaram que os genes do ciclo celular regulados por RB/E2F estavam associados a PFS curto na coorte combinada (Fig. 8a, b ), bem como em cada um dos grupos de tratamento individuais (Fig. 8c ). Para explorar as características preditivas desses conjuntos de genes, utilizamos genes comuns do ciclo celular nos 100 principais genes associados à FC nas coortes AI e FUL. Esta lista de 10 genes foi significativamente associada à PFS tanto nas coortes AI quanto nas coortes FUL (Fig. 8d-g ). No entanto, este conjunto de genes combinados foi inferior aos principais genes do ciclo celular seletivos para AI ou FUL (Fig. 8h-k ).
a Genes associados a PFS mais curta, conforme identificado pela regressão univariada de riscos proporcionais de Cox em cada uma das coortes de tratamento. −Log 10 valores p da coorte AI e FUL são mostrados no eixo x e no eixo y , respectivamente. Os dados são filtrados por HR < 1, valor de p ≤ 0,1 (coorte AI) ou valor de p ≤ 0,05 (coorte fulvestrant). b , c Análise de enriquecimento de conjunto de genes para genes associados a PFS mais curtos. Os valores de HR da análise univariada de Cox PH nas coortes combinadas foram utilizados para realizar a análise pré-classificada do GSEA. Os valores de HR são subtraídos por 1 para centralizar os genes sem associação em zero. Gráficos d – g Heatmap e KM para módulos genéticos comuns do ciclo celular testados na coorte tratada com AI ( d , e ) ou FUL ( f , g ). p = 0,13 e p = 0,01, respectivamente, pelo teste log rank. h – k Teste de módulos genéticos do ciclo celular identificados em cada coorte separadamente para associação com PFS na mesma coorte. p = 0,003 e p < 0,001, respectivamente, pelo teste log rank. As colunas nesses mapas de calor são ordenadas por valores médios de expressão gênica normalizada transformada em log2, de alto (esquerda) a baixo (direita).
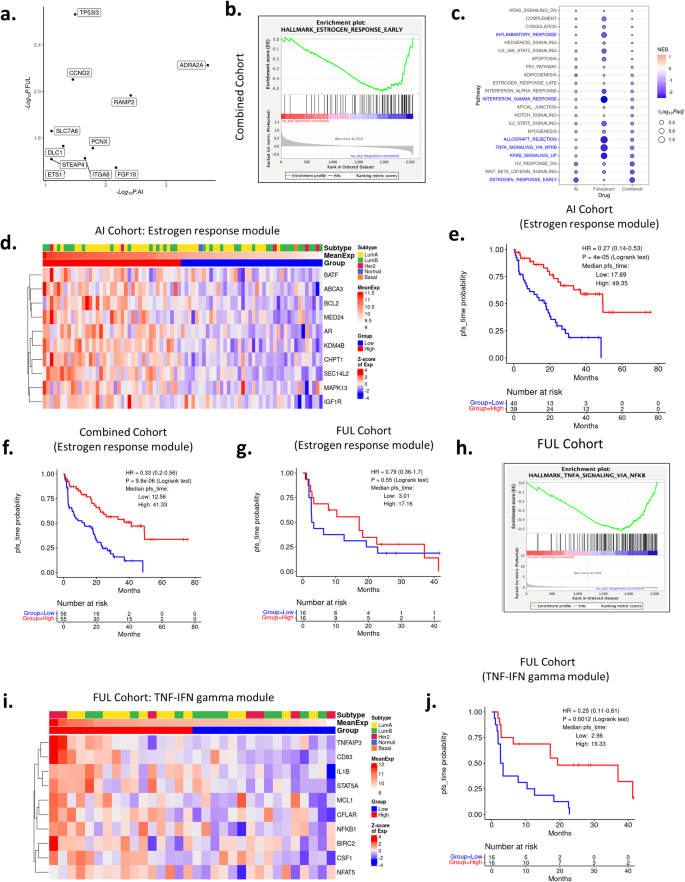
Em termos de PFS mais longo, foram observados menos genes comuns para tratamento com IA e FUL para associação (Fig. 9a ). Pelas análises GSEA, a sinalização do receptor de estrogênio foi o principal conjunto de genes associado a PFS mais longo na coorte combinada e no grupo de inibidores AI + CDK4/6 (Fig. 9b, c ). Um módulo de 10 genes associados à sinalização do receptor de estrogênio foi fortemente associado à PFS na coorte AI (Fig. 9d, e ) e na coorte combinada (Fig. 9f ). No grupo inibidor FUL + CDK4/6, esta via apenas tendeu a uma PFS mais longa (Fig. 9g ). Análises do grupo FUL + CDK4/6 usando a mesma metodologia mostraram que os conjuntos de genes de sinalização TNF e interferon-gama (IFN gama) estavam associados a PFS mais longos (Fig. 9c, h ). Esta associação foi confirmada no contexto de agrupamento baseado na distância euclidiana e subsequentes análises de KM (Fig. 9i, j ). Assim, embora as assinaturas do ciclo celular sejam comumente associadas a PFS mais curtos, existem programas diferenciais que parecem dominantes para AI versus FUL para PFS mais longos.
a Genes associados a PFS mais longa, conforme identificado pela regressão univariada de riscos proporcionais de Cox em cada uma das coortes de tratamento. −Log 10 valores p da coorte AI e FUL são mostrados no eixo x e no eixo y , respectivamente. Os dados são filtrados por HR < 1, valor de p ≤ 0,1 (coorte AI) ou valor de p ≤ 0,05 (coorte fulvestrant). b O conjunto precoce de genes da resposta estrogênica está associado a uma PFS mais longa, conforme revelado pela GSEA na coorte combinada. c Gráfico de bolhas mostrando enriquecimento diferencial para conjuntos de genes entre coortes tratadas com IA e fulvestrant. d Mapa de calor usando genes do módulo de resposta ao estrogênio em dados de expressão gênica de biópsias pré-tratamento na coorte tratada com IA. O gráfico KM mostrando a diferença de PFS entre os valores médios de expressão altos e baixos deste módulo do gene de resposta ao estrogênio na coorte tratada com IA. p < 0,001 pelo teste de log rank. Gráficos f , g KM mostrando diferença de sobrevivência entre grupos de expressão gênica média baixa e alta do módulo genético de resposta ao estrogênio nas coortes tratadas combinadas ( f ) ou fulvestrant ( g ). p < 0,001 e p = 0,55, respectivamente, pelo teste log rank. h Gráfico GSEA mostrando enriquecimento de genes na sinalização de TNF via conjunto de genes NFKB. i , j Heatmap e gráfico KM mostrando o padrão de expressão gênica do módulo do gene TNF-IFN gama e seu poder na estratificação de pacientes na coorte tratada com fulvestrant usando dados de expressão gênica das biópsias pré-tratamento. p = 0,0012 pelo teste de log rank.
Discussão
Aqui usamos dados clínicos e amostras de tecidos do “mundo real” para explorar características de expressão gênica de resposta à terapia padrão baseada em inibidores de CDK4/6 no câncer de mama metastático HR +/HER2−. Até o momento, o único marcador empregado na administração de inibidores de CDK4/6 nesta população é o status de ER e HER2, que é usado para direcionar a terapia endócrina, embora características potenciais de resposta tenham sido descritas a partir das análises dos ensaios clínicos randomizados 26 , 38 , 39 , 40 .
Como além do RE, o status da RP era conhecido, pudemos avaliar como o status da RP se associa à duração da PFS. Em nossa coorte, composta por pacientes tratados em grande parte com palbociclib e IA ou FUL, o status de RP foi relevante na coorte de tratamento com IA. Nosso achado é consistente com outro estudo publicado recentemente no mundo real que avaliou a RP em uma coorte menor de pacientes 50 . Semelhante às nossas observações, eles descobriram que a RP não foi significativa na coorte tratada com FUL, sugerindo que as distinções biológicas relacionadas à sensibilidade endócrina e aos tratamentos anteriores terão um impacto nos supostos biomarcadores. A pontuação de Nottingham (pontuação SBR modificada) representa um composto de características patológicas associadas à diferenciação tubular, pleomorfismo nuclear e taxa mitótica. Conforme mostrado aqui, os escores do SBR foram associados à duração da PFS em nossa coorte. Tal como acontece com o PR, o SBR mostrou significância apenas nos grupos tratados com IA. Estas descobertas e o trabalho realizado com PALOMA-2 e PALOMA-3 39 sugerem que os biomarcadores desenvolvidos provavelmente terão que ser seletivos para a linha de tratamento. Curiosamente, um marcador robusto para a duração da PFS na coorte de IA foi desenvolvido simplesmente combinando o status SBR e PR. No braço com SBR3 e PR baixa/deficiente, a PFS foi de 6,21 meses, sugerindo que as estimativas da provável PFS poderiam ser deduzidas a partir de um número relativamente pequeno de medidas patológicas existentes em doentes tratados com inibidor de CDK4/6 e IA.
Para ir além dos marcadores clínicos comuns, utilizamos o painel de biomarcadores HTG Oncology, que tem sido empregado em múltiplas análises de ensaios clínicos randomizados 23 , 26 , 38 . Utilizando subtipagem molecular (AIMS), definimos um grau de plasticidade na expressão gênica durante a terapia. Tanto na recorrência remota quanto no cenário metastático antes do tratamento combinado com inibidor de CDK4/6, houve frequências comparáveis de tumores luminais A e luminais B. Durante o tratamento, há uma mudança para os subtipos luminal A e normal. Este perfil de resposta é ostensivamente devido ao efeito no alvo dos inibidores de CDK4/6 na supressão de genes do ciclo celular que denotam a diferença predominante do luminal B em oposição ao luminal A e aos subtipos normais. Curiosamente, a investigação do comportamento de tumores que “mudaram” para o luminal A em vez de se originarem do luminal A revelou que o luminal A antes do tratamento tinha uma PFS mais longa, embora os números sejam muito limitados para fazer uma conclusão robusta (Figura 8 suplementar). ). Após a progressão da doença, poucos tumores luminais A permaneceram e houve uma desregulação geral adicional dos genes do ciclo celular. Estes dados sugerem que embora a terapia seja eficaz na supressão da proliferação, uma vez que os tumores progridem, eles evoluíram para uma biologia mais agressiva. Isto também pode explicar por que nesta coorte, a OS pós-progressão é essencialmente idêntica. No contexto do presente estudo, o subtipo de doença na biópsia pré-tratamento foi associado à duração da SLP. Isto seguiu em grande parte o significado prognóstico dos subtipos de câncer de mama em que luminal B, Her2 e basal estão associados a resultados desfavoráveis. Curiosamente, esta associação de subtipos só foi significativa no subgrupo tratado com IA. Parte da falta de significância pode ser devida ao número relativamente pequeno de pacientes na coorte tratada com FUL, bem como à maior heterogeneidade em relação aos subtipos além dos luminais A e B.
Para abordar as características biológicas associadas à PFS, foi empregada uma abordagem relativamente imparcial. Curiosamente, mostrou que a desregulação do ciclo celular, exemplificada pelos genes alvo E2F ou genes associados a G2/M, está conjuntamente associada à PFS encurtada, independentemente da terapia endócrina utilizada, especificamente quando aplicada à biópsia pré-tratamento (Figura 9 suplementar ). Esses achados são semelhantes aos emergentes das análises do PALOMA-2/3 26 , 38 , 39 , 40 . No entanto, ao nível do gene, houve diferenças entre as coortes de tratamento. Da mesma forma, parece que a expressão de CDK4/6 é relevante no contexto de tumores tratados com IA, enquanto CDK2 e CCNE1 são mais relevantes no contexto de tumores tratados com FUL. Assim, permanece desconhecido se a importância de genes alvo E2F selecionados é devida a um mecanismo primário de resistência, por exemplo, desregulação de CCNE1 vs CDK6. Em termos de PFS mais longa, a sinalização do receptor de estrogênio foi fortemente associada à duração livre de progressão, mais potentemente na biópsia pré-tratamento em oposição à amostra primária/recidiva (Figura 9 suplementar ). Esse achado também foi observado nas análises dos estudos PALOMA-2/3 39 . No entanto, na nossa coorte tratada com FUL houve enriquecimento mínimo para estes genes associados à duração da PFS. Em contraste, os conjuntos de genes TNF e IFN-gama foram mais fortemente associados a uma PFS mais longa em coortes tratadas com FUL. Isto também pode estar relacionado à associação de CCND2 com a duração da PFS, predominantemente no grupo de tratamento FUL 47 . Este achado contrasta com outros cenários, onde foi proposto que os genes associados à sinalização do interferon estão associados à resistência aos inibidores de CDK4/6 em populações clínicas 26 . Os conjuntos de genes TNF e IFN-gama são altamente heterogêneos e será importante avaliar genes individuais entre grupos experimentais para, em última análise, chegar a conclusões fortes. Acreditamos que pelo menos parte do sinal detectado na coorte FUL reflete os TILs que geralmente estão associados a um melhor prognóstico no câncer de mama HR+/HER2−. Genes imunologicamente restritos (por exemplo, CD40 e CD3D) foram seletivamente associados a PFS mais longa na coorte tratada com FUL (Figura 8 suplementar ). Curiosamente, usando dados METABRIC 51 , poderíamos comparar a associação dos conjuntos de genes aqui empregados com o prognóstico (Figura 10 Complementar ). Os módulos do ciclo celular estão associados ao prognóstico, no entanto, nem os módulos do gene estrogênio nem do TNF foram associados à sobrevida livre de doença em amostras METABRIC. Surpreendentemente, apenas o módulo de resposta ao estrogênio está fortemente associado à OS nesta coorte (Figura 11 Complementar).
No total, os estudos aqui identificaram múltiplas características biológicas que emergem durante o tratamento com inibidores de CDK4/6 e sugeriram vias relevantes para a PFS. No entanto, mais estudos serão necessários para desenvolver esses genes ou características clínicas até o ponto de um biomarcador. Estudos em andamento estão desenvolvendo coortes independentes para validar os resultados aqui e para desenvolver biomarcadores validados prospectivamente para a duração da resposta ao CDK4/6 com regimes de terapia endócrina.
Métodos
Fonte de dados e seleção de pacientes
A revisão de prontuários foi realizada para 280 pacientes que foram diagnosticados com câncer de mama HR+/HER2− e receberam um inibidor de CDK4/6 de 2015 a 2022 no Roswell Park Comprehensive Cancer Center como critério de inclusão para o estudo. A grande maioria (>95%) dos pacientes consentidos eram mulheres. Foram utilizados dados de pacientes de dois estudos aprovados pelo Conselho de Revisão Institucional do Roswell Park Comprehensive Cancer Center. Um protocolo retrospectivo foi utilizado para coletar informações e bioespécimes em 71 pacientes. Posteriormente, foi desenvolvido um protocolo combinado retrospectivo e prospectivo (NCT04526587). Duzentos e vinte e nove pacientes foram consentidos em 22 de dezembro de 2022. Os pacientes elegíveis tinham ≥18 anos de idade, tinham câncer de mama avançado ER+/HER2− e foram tratados com um inibidor de CDK4/6. Registros médicos eletrônicos foram usados para extrair informações demográficas, histórico de tabagismo, estado da menopausa, IMC, status de desempenho do Eastern Cooperative Oncology Group (ECOG), relatórios de cirurgia e patologia, dados genômicos, datas de diagnóstico e recorrência(s), local(is) de metástases e informações sobre tratamento do câncer.
Ética
As informações clínicas coletadas foram aprovadas pelo Conselho de Pesquisa Institucional do Roswell Park Cancer Center. O consentimento informado por escrito foi obtido para todos os pacientes por meio do ensaio clínico (NCT04526587) ou do protocolo Roswell Park Remnant Tissue e do protocolo correspondente do investigador aprovado pelo IRB específico para este estudo.
Seleção de tecido
Todos os slides de casos de patologia cirúrgica do Departamento de Patologia do Roswell Park Comprehensive Cancer Center foram revisados para cada paciente para garantir tecido tumoral adequado. Os casos escolhidos foram então codificados por momentos específicos durante o seu tratamento, como mostrado na Fig . Os blocos selecionados foram seccionados e as regiões de alta celularidade tumoral foram definidas por um patologista mamário (AKW). As lâminas foram então enviadas para a HTG Molecular Diagnostics, Inc. para expressão genética direcionada consistente com outros estudos 26 , 39 , 40 .
Análise estatística
A PFS foi definida como o tempo desde a primeira dose da terapia com inibidor de CDK4/6i até a progressão comprovada por exame ou marcador, ou morte durante a terapia. A OS foi definida como o momento da biópsia (ou exame se nenhuma biópsia foi realizada) que levou ao início da terapia com inibidor de CDK4/6, até a morte ou a data do último acompanhamento. Os pacientes que interromperam a terapia com ciclib devido a toxicidades ( n = 22) não foram considerados nos cálculos da PFS. Para o objetivo principal deste estudo, os pacientes foram divididos em 2 grupos: pacientes que tomavam um inibidor da aromatase (letrozol, anastrozol ou exemestano) com um inibidor de CDK 4/6 e pacientes que tomavam fulvestrant com um inibidor de CDK 4/6. A análise de sobrevivência KM comparada com testes log-rank, regressão univariada e multivariada de riscos proporcionais de Cox foi usada para comparar a PFS por terapia endócrina. R versão 3.6.1 ou 4.2.0 (R Foundation for Statistical Computing, Viena, Áustria) foi utilizada para todas as análises estatísticas.
Preparação e processamento de dados RNA-Seq
Amostras de tecido de pacientes obtidas durante o tratamento padrão de vários momentos clínicos foram enviadas à HTG Molecular Diagnostics, Inc. para sequenciamento direcionado usando seu Painel de Biomarcadores Oncológicos HTG EdgeSeq que consiste em 2.549 genes selecionados associados ao câncer. Os dados brutos foram reunidos a partir de quatro execuções separadas de HTG. As contagens de leitura bruta de quatro lotes foram submetidas à remoção do efeito de lote usando ComBat-seq 52 . A matriz de contagem de leitura bruta corrigida pelo efeito de lote é então usada como entrada para edgeR 53 para normalização de dados. Os dados normalizados foram então transformados em log2 adicionando uma pseudo-contagem de 1 a cada valor na matriz de dados e posteriormente usados para análises downstream.
Análise de expressão gênica diferencial de amostra pareada
A análise diferencial da expressão gênica foi realizada entre diferentes momentos (em tratamento vs pré-tratamento, pós-tratamento vs pré-tratamento, pré-tratamento vs primário/recorrência) em amostras de pacientes pareadas usando o pacote DESeq2 54 (v1.36.0) Bioconductor.
Análise de Enriquecimento de Conjunto Genético (GSEA)
Usamos um software GSEA instalado localmente (v4.2.1) para realizar análises pré-classificadas do GSEA para identificar associações de conjuntos de genes no conjunto de dados HALLMARK no banco de dados de assinatura molecular (h.all.v2023) com listas de genes classificadas por taxas de risco (subtraídas por um , para centralizar o HR = 1 (sem associação) em zero) (Figs. 8b e 9b, h ) ou com magnitude (Log2FC) de genes diferencialmente expressos entre pontos no tempo (Fig. 4d ). Para obter pontuação de enriquecimento normalizada e valores de p para gerar gráficos nas Figs. 4c , 8c e 9c , usamos o pacote fgsea Bioconductor ( https://doi.org/10.1101/060012 ) e código R personalizado.
Conjuntos de dados externos usados neste estudo
O conjunto de dados NeoPalAna foi recuperado do Gene Expression Omnibus (GEO) com número de acesso GSE93204. Utilizamos o pacote GEOquery (v2.64.2) Bioconductor para recuperar os dados de expressão gênica e informações clínicas. O conjunto de dados METABRIC 51 foi baixado do cBioPortal. Para corresponder às características dos pacientes, extraímos um subconjunto deste conjunto de dados para incluir apenas os pacientes com status HR+/HER2−.
Atribuição de subtipo intrínseco
Para prever subtipos intrínsecos de câncer, usamos o pacote AIMS 45 R. Como o painel do gene HTG não inclui todos os genes usados no AIMS, avaliamos a precisão da classificação do subtipo com o conjunto reduzido de genes usado no painel HTG. Primeiro executamos o código de exemplo na documentação do AIMS com os dados incluídos nesse pacote. Em seguida, executamos o código novamente com o conjunto de dados de teste reduzido, incluindo apenas genes disponíveis no painel de genes HTG. Isto resultou na classificação incorreta de 52 amostras de um total de 321 amostras (84% de concordância). Para executar o AIMS, substituímos o símbolo do gene na matriz de dados de contagem de leitura de RNA-Seq bruto pelo ID do gene Entrez usando o pacote org.Hs.eg.db Bioconductor R (v3.10.0).
Seleção de módulo genético
A análise de regressão univariada de Cox foi realizada em cada uma das coortes de tratamento (IA, fulvestrant e combinado) separadamente, usando os dados de expressão gênica de biópsias pré-tratamento. Os valores da razão de risco foram subtraídos por 1 para obter valores zero indicando nenhuma associação (HR = 1) e usados na análise pré-classificada do GSEA em relação aos conjuntos de genes HALLMARK (h.all.v2023). Identificamos os alvos E2F e o ponto de verificação G2M como os conjuntos de genes mais enriquecidos, a resposta de estrogênio precoce como o conjunto de genes mais esgotados na coorte tratada com IA, a resposta de IFN-gama e a sinalização de TNF via NFKB como os conjuntos de genes mais esgotados na coorte tratada com fulvestrant . Os genes de ponta em cada um dos conjuntos de genes mais enriquecidos/esgotados são cruzados com os 100 principais genes de alta FC ou com os 100 genes de baixa FC mais baixos. Traçamos mapas de calor para esses conjuntos de genes e selecionamos dez genes com base em seu padrão de expressão. Nós os denominamos como “Módulo de Resposta de Estrogênio” para os 10 genes selecionados usando a coorte tratada com IA, “Módulo TNF-IFN gama” para os 10 genes selecionados usando a coorte tratada com fulvestrant, e “Módulo de Ciclo Celular Comum” para os dez genes selecionados das coortes combinadas de IA e fulvestrant.
Plotagem
O gráfico Sankey foi feito usando o pacote plotly R (v4.9). Primeiro realizamos a previsão de subtipos intrínsecos de tumor para todas as amostras usando AIMS. Os resultados de previsão para amostras do mesmo paciente com pontos de tempo diferentes (primário, pré-tratamento, durante o tratamento e pós-tratamento) foram usados como entrada para um script R personalizado para criar o gráfico de Sankey. Todos os mapas de calor são agrupados usando o pacote ComplexHeatmap 55 R (v2.12.1). Os gráficos KM são baseados no pacote Survival R (3.4–0) com código wrapper personalizado para melhorias. Outras parcelas foram preparadas usando os pacotes R ggplot2 (v3.3.6) ou ggpubr (v0.4.0).
Coloração mIF
Secções de 4 µm fixadas em formalina e embebidas em parafina (FFPE) foram cortadas e colocadas em lâminas carregadas. As lâminas foram secas a 65°C por 2 horas. Após a secagem, as lâminas foram colocadas no BOND RX m Research Stainer (Leica Biosystems) e desparafinadas com solução BOND Dewax (AR9222, Lecia Biosystems). O processo de coloração imunofluorescente multiespectral (mIF) envolveu repetições em série do seguinte para cada biomarcador: recuperação/remoção de epítopo com ER1 (tampão citrato pH 6, AR996, Leica Biosystems) ou ER2 (tampão Tris-EDTA pH9, AR9640, Leica Biosystems), tampão de bloqueio (AKOYA Biosciences), anticorpo primário, anticorpo secundário Opal Polymer HRP (AKOYA Biosciences), Opal Fluorophore (AKOYA Biosciences). Todos os reagentes AKOYA usados para coloração mIF vêm como um kit (NEL821001KT). Spectral DAPI (AKOYA Biosciences) foi aplicado assim que as lâminas foram removidas do BOND. Eles foram cobertos usando um método aquoso e meio de montagem antidesbotamento Diamond (Invitrogen ThermoFisher). O painel mIF consistiu nos seguintes anticorpos: Ki67 (Abcam, ab16667), AE1AE3 (Dako, M3515), CCNE (Abcam, ab33911), CCND1 (ThermoFisher, MA1-39546), CCNA (Abcam, ab32386), RB (Sinalização Celular , 9309s), pRB (sinalização celular, 8516) e MCM2 (BioSb, BSB6334).
Imagem e análise de tecidos
Os slides foram fotografados no PhenoImager™ HT (AKOYA Biosciences). Análises adicionais das lâminas foram realizadas utilizando o software inForm® v2.6.0 (AKOYA Biosciences). As lâminas inteiras foram primeiro digitalizadas em uma visualização não misturada e, em seguida, ROIs representativos foram selecionados para aquisição sob a orientação de um patologista. Essas ROIs foram então digitalizadas novamente para obter a separação espectral completa. Um subconjunto representativo desses ROIs não misturados foi então usado para treinar a segmentação de tecidos e células. Em seguida, foi criado um algoritmo exclusivo usando uma técnica de aprendizado de máquina, na qual o operador seleciona exemplos de células positivas e negativas para cada marcador. Esses algoritmos foram então aplicados em lote em um número maior de ROIs selecionados para inclusão em análises posteriores. O plugin RStudio, phenoptrReports, foi usado para extrair contagens de fenótipos das tabelas de dados resultantes.




Comentários
Postar um comentário